2. 青岛大学医学院附属烟台毓璜顶医院耳鼻咽喉头颈外科, 山东 烟台 264000
2. Department of Otolaryngology & Head and Neck Surgery, Yantai Yuhuangding Hospital, Medical College of Qingdao University, Yantai 264000, Shandong, China
甲状腺癌是内分泌系统中最常见的恶性肿瘤,其发病率在头颈部恶性肿瘤中居首位,占全身恶性肿瘤的3%[1, 2]。其组织学类型主要包括乳头状癌、滤泡状癌、未分化癌和髓样癌,以乳头状甲状腺癌(papillary thyroid cancer,PTC)所占比例最大,约占80%[3]。近年来,甲状腺癌的发病率在全球以年均5%的速度增长,我国的甲状腺癌发病率由1/10万上升到3~4/10万[4]。目前高发病率造成了人群一定程度的心理恐慌,特别是位于沿海城市的居民由于碘的摄入量要远大于内陆地区,他们对甲状腺疾病的关注更是不断提高。甲状腺癌的病因学迄今为止仍是环境和遗传两种因素,但其内在的发病机制仍不十分明确,目前基于甲状腺癌病因学的预防治疗研究较少,直接影响甲状腺癌的临床预防。我们现就DSB修复基因的多态性与甲状腺癌相关性的研究做一综述。
1 甲状腺癌与DNA双链断裂DSB(double-strand break,DSB)目前惟一明确的甲状腺癌外源性致病因素是电离辐射,在暴露于切尔诺贝利核泄露的群体中,甲状腺癌增加了62倍[5]。而电离辐射对DNA的主要损伤是引起DNA双链断裂,也是DNA损伤中最为致命的一种。当细胞的DNA双链断裂修复能力出现异常,细胞内遗传信息将不能正确而完整的传递给子代,终将导致细胞的死亡或突变体的产生,而后者正是疾病发生的一个重要原因。异常修复将会导致染色体的易位、基因重排和染色体畸形等基因突变。上述基因改变直接影响基因组的稳定性,最终导致细胞的癌变。因此对细胞而言拥有健全、快速修复DSB的机制是至关重要的[2]。
2 HR与NHEJ修复机制在哺乳动物中,DSB的修复主要有两种机制:同源重组(HR)和非同源末端连接(NHEJ)[6]。受同源染色体的影响,HR修复通路主要限于在哺乳动物中S后期和G2期[7]。NHEJ修复过程由于不需要姐妹染色单体的同源序列作为模板,从而活跃贯穿整个细胞周期,上述修复途径在DSB修复过程中互为补充[8]。但是NHEJ拥有可在任意细胞周期起作用这一优势,其发生频率是HR 的100至1 000倍,是DSB修复的主要途径[9, 10, 11]。
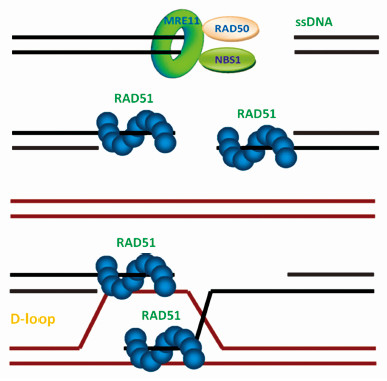
HR修复的主要成分为MRN(MRE11-RAD50-NBS1)复合体。DNA修复相关蛋白51(DNA damage repair protein 51 RAD51)是该通路的核心,其同源物主要包括:RAD51B、RAD51C、RAD51D、XRCC2和XRCC3。HR修复过程大致分为联合前期、联合期与联合后期。
联合前期:MRN复合体与DNA断端结合并持续产生ssDNA(single stranded DNA),继而RAD51在ssDNA上形成联合前核蛋白纤维;
联合期:RAD51-ssDNA联合前核蛋白纤维介导同源序列的寻找和DNA链侵入。靶DNA与RAD51核蛋白纤维之间的DNA链配对形成D-loop,并以DNA同源序列为模板启动DNA修复的过程;
联合后期:以3′-断端为引物进行DNA合成。HR修复机制示意图,见图 1。
|
图1 HR修复机制示意图 Fig. 1 The schematic of repair mechanism of HR |
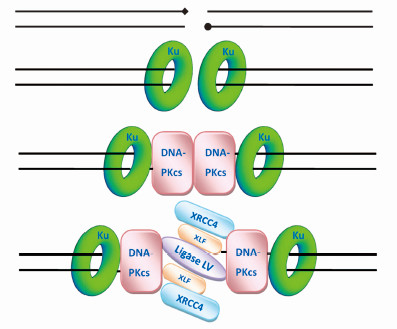
NHEJ路径的核心分子有DNA-PK、X-射线修复交叉互补蛋白4(X-ray repair cross complementing 4,XRCC4)、DNA连接酶IV(DNA lignaseIV,LigIV)等,DNA-PK是由催化亚基DNA-PKcs(the DNA-dependent protein kinase catalytic subunit)和Ku70、Ku80组成的全酶,其中XRCC5编码Ku80蛋白、XRCC6 编码Ku70 蛋白、XRCC7编码DNA依赖型蛋白激酶催化亚基(DNA-PKcs)蛋白[12, 13]。NHEJ 的基本过程是:Ku蛋白识别断裂末端并最先与断裂DNA 结合,随后Ku招募DNA-PKcs在空间上排列并稳定两个断裂末端,最后连接酶LigIV -XRCC4复合体执行连接功能。NHEJ修复机制示意图,见图 2。
|
图2 NHEJ修复机制示意图 Fig. 2 The schematic of repair mechanism of NHEJ |
DNA损伤修复基因泛指那些编码产物在功能上参与DNA损伤识别和修复的基因,这类基因的突变会使损伤DNA得不到及时有效的修复,或发生错误修复,积累至一定程度就可导致细胞死亡或癌变[14, 15]。修复基因的单核苷酸多态性(single nucleotide polymorphisms,SNPs)作为DNA多态性的一种,是指染色体基因组水平上单个核苷酸变异引起的DNA序列变化,被认为是人们疾病易感性和药物反应的决定性因素。因此,对于修复基因SNPs的研究已成为当前探究肿瘤发病机制的热点领域[16]。
近年来,针对DNA损伤修复基因的研究逐渐增多,但是涉及到与甲状腺癌易感性相关的DNA损伤修复基因的却很少。针对这一科研课题,2009年 Bastos等[17]对以葡萄牙语为母语的白人群体中的109例甲状腺癌患者和217位健康人利用病例——对照分析方法进行了一项关于同源重组修复基因多态性与甲状腺癌易感性的研究,研究结果表明在XRCC3 T241M,XRCC2 R188H,NBS1 E185Q,RAD51 Ex1-59G>T这4个修复基因多态性中XRCC3 T241M(OR=2.0;95% CI=1.1-3.6;P=0.026)与RAD51 5′UTR(Ex1-59G>T)(OR=1.9;95% CI=1.0-3.5;P=0.057)这两个SNP位点增加个体对甲状腺癌的易感性。 Gomes等[18]于2010年利用同样的方法对同一样本再次针对非同源末端重组修复基因的多态性与甲状腺癌易感性进行了研究,在他们对2个XRCC4 (Asn298Ser,Thr134Ile),1个LIG4 (Thr9Ile),4个Ku80 3′UTR(Ex21-238G>A,Ex21+338T>C,Ex21-352C>A,Ex21+466A>G),共计7个SNPs进行了基因型分析。他们的研究表明,Ku80 Ex21-238G>A,Ex21+338T>C多态性可能影响个体对甲状腺癌的易感性(OR=2.281;95% CI=1.063-4.894;P=0.034)。众多研究表明,Ku80多态性对肿瘤易感性的影响涉及乳腺癌、膀胱癌、神经胶质瘤、骨髓瘤及小细胞肺癌[19, 20, 21, 22, 23, 24]等多种恶性肿瘤。在同源重组修复中,对于XRCC1与XRCC3的多态性研究较多。其中XRCC1C18067T(Thr241Met)多态性对甲状腺癌的易感性研究结果较为一致: Bastos等[17]对该SNP的研究表明XRCC3 T241M(OR=2.0;95% CI=1.1-3.6;P=0.026;Stugris等[25]对134例美国白人分化型甲状腺癌患者(DTC)及其对照的研究表明:XRCC3基因C18067T(Thr241Met)多态性与DTC的发病相关,OR=2.1;针对这一突变,Yuan等[26]对汉族人群进行了分析,结果表明与Stugris等[25]研究相似,C18067T(Thr241Met)多态性显著改变了PTC易感性,OR=2.93。在对XRCC1基因的单核苷酸多态性研究中,对Arg194Trp、Arg280His和Arg399Gln的多态性研究较为广泛[27]。但是此项研究结果众说纷纭,有时甚至自相矛盾。针对这种现状Bao等[28]在2013年对这三个基因的多态性做了Meta分析,结果显示Arg280His基因的多态性增加了白人种群对分化型甲状腺癌的易感性,但是,对于亚洲人群而言,却能起到抵御甲状腺癌的作用。Ryu等[29]针对韩国人群中XRCC1Arg194Trp突变进行了相关研究,结果表明Arg/Trp基因型明显降低了甲状腺乳头状癌的发病风险(OR=0.55)。同样是XRCC1Arg194Trp这个SNP位点,Chiang[30]等在台湾地区进行了一次病例对照研究,结果却显示携带基因型Trp/Trp者患甲状腺癌的风险明显增高(OR=1.85)。此外,针对XRCC1 Arg399Gln突变,Zhu等[31]研究结果表明XRCC1第399密码子基因型Gln/Gln可能是PTC的易感基因型(OR=4.65)。相对XRCC1、XRCC3对甲状腺癌易感性的研究,关于XRCC7对甲状腺癌的研究较为稀少,2012年Rahimi等[32]发表了第一篇关于XRCC7与甲状腺癌易感性相关的文章,该研究针对伊朗人群Ile3434Thr这一基因多态性对173例甲状腺癌及204名无瘤者进行了一组临床病例-对照试验研究,结果显示携带这一基因型的个体患甲状腺癌的风险明显升高(OR:2.42,95% CI=1.55-3.81,P=0.000 1)。直观反映相关修复基因SNP与甲状腺癌发病风险的关系,见表 1。
| 表1 修复基因SNP与甲状腺癌易感性关联研究汇总 Tab. 1 A summary of the association between DSB repair gene polymorphism and susceptibility to thyroid carcinoma |
以上研究在一定程度上证实了DNA损伤修复基因多态性对基因组稳定性有深远影响,修复基因的单核苷酸多态性与甲状腺癌疾病遗传易感性相关。
4 总结与展望DNA修复机制通过逆转DNA损伤进而维护DNA基因组的完整性和稳定性。而DNA修复基因的遗传变异通过改变DNA修复能力进而改变癌症的发病风险。
人类基因序列的变异大多数是单核苷酸的突变,而SNP在不同的人群中的分布存在差异,这些差异可以代表某一种族或某人群间的遗传变异。因此,研究SNP有助于解释个体的表型差异、不同群体和个体癌性疾病发病的差异。通过筛选与甲状腺癌易感性显著相关的SNP基因或单体型,作为分子遗传标志,进行分子标志辅助选择和分子诊断,通过流行病学调查,确定患甲状腺癌的高危人群,进而可以进行患病危险程度的评估,建立有效的预警系统,达到有效的预防目的。另外,亦有研究指出,通过对基因SNP与甲状腺癌易感性的相关性研究,确定甲状腺癌相关的遗传易感基因,探寻甲状腺癌的基因治疗的靶点,这将为甲状腺癌研究开创新的领域,为甲状腺癌患者带来福音。
| [1] | Siraj A K, Hussain A R, Al-Rasheed M, et al. Demethylation of TMS1 gene sensitizes thyroid cancer cells to TRAIL-induced apoptosis[J]. J Clin Endocrinol Metab, 2011, 96(1):E215-224.( 1) 1) |
| [2] | Schneider D F, Chen H. New developments in the diagnosis and treatment of thyroid cancer[J]. CA Cancer J Clin, 2013, 63(6):373-394.( 1) 1) |
| [3] | Hundahl S A, Fleming I D, Fremgen A M, et al. A National Cancer Data Base report on 53, 856 cases of thyroid carcinoma treated in the U.S., 1985-1995[J]. Cancer, 1998, 8(12):2638-2648.( 1) 1) |
| [4] | 钱碧云, 陈可欣, 何敏, 等. 天津市区甲状腺癌流行状况调查[J]. 中国肿瘤临床, 2005, 3294):218-221.( 1) 1) |
| [5] | Knauf J A, Ward L S, Nikiforov Y E, et al. Isozyme-specific abnormalities of PKC in thyroid cancer: evidence for post-transcriptional changes in PKC epsilon[J]. J Clin Endocrinol Metab, 2002, 87(5):2150-2159.( 1) 1) |
| [6] | Hartlerode A J, Scully R. Mechanisms of double-strand break repair in somatic mammalian cells[J]. Biochem J, 2009, 423(2):157-168.( 1) 1) |
| [7] | Symington L S, Gautier J. Double-strand break end resection and repair pathway choice[J]. Annu Rev Genet, 2011, 45:247-271.( 1) 1) |
| [8] | Shrivastav M, De Haro L P, Nickoloff J A. Regulation of DNA double-strand break repair pathway choice[J]. Cell Res, 2008, 18(1):134-147.( 1) 1) |
| [9] | Lieber M R, Ma Y, Pannicke U, et al. Mechanism and regulation of human non-homologous DNA end-joining[J]. Nat Rev Mol Cell Biol, 2003, 4(9):712-720.( 1) 1) |
| [10] | Zhang H, Hasty P, Bradley A. Targeting frequency for deletion vectors in embryonic stem cells[J]. Mol Cell Biol, 1994, 14(4):2404-2410.( 1) 1) |
| [11] | Burma S, Chen B P, Chen D J. Role of non-homologous end joining (NHEJ) in maintaining genomic integrity[J]. DNA Repair (Amst), 2006, 5(9-10):1042-1048.( 1) 1) |
| [12] | Summers K C, Shen F, Sierra Potchanant E A, et al. Phosphorylation: the molecular switch of double-strand break repair[J]. Int J Proteomics, 2011, 2011(2011):373816.( 1) 1) |
| [13] | Tolle R. Information technology tools for efficient SNP studies[J]. Am J Pharmacogenomics, 2001, 1(4):303-314.( 1) 1) |
| [14] | Sehl M E, Langer L R, Papp J C, et al. Associations between single nucleotide polymorphisms in double-stranded DNA repair pathway genes and familial breast cancer[J]. Clin Cancer Res, 2009, 15(6):2192-2203.( 1) 1) |
| [15] | Walsh T, King M C. Ten genes for inherited breast cancer[J]. Cancer Cell, 2007, 11(2):103-105.( 1) 1) |
| [16] | Fornage M, Doris P A. Single-nucleotide polymorphism genotyping for disease association studies[J]. Methods Mol Med, 2005, 108:159-172.( 1) 1) |
| [17] | Bastos H N, Anto M R, Silva S N, et al. Association of polymorphisms in genes of the homologous recombination DNA repair pathway and thyroid cancer risk[J]. Thyroid, 2009, 19(10):1067-1075.( 1) 1) |
| [18] | Gomes B C, Silva S N, Azevedo A P, et al. The role of common variants of non-homologous end-joining repair genes XRCC4, LIG4 and Ku80 in thyroid cancer risk[J]. Oncol Rep, 2010, 24(4):1079-1085.( 1) 1) |
| [19] | Kuschel B, Auranen A, McBride S, et al. Variants in DNA double-strand break repair genes and breast cancer susceptibility[J]. HumMol Genet, 2002, 11(12):1399-1407.( 1) 1) |
| [20] | Figueroa J D, Malats N, Rothman N, et al. Evaluation of genetic variation in the double-strand break repair pathway and bladder cancer risk[J]. Carcinogenesis, 2007, 28(8):1788-1793.( 1) 1) |
| [21] | Roddam P L, Rollinson S, ODriscoll M, et al. Genetic variants of NHEJ DNA ligase IV can affect the risk of developing multiple myeloma, a tumour characterized by aberrant class switch recombination[J]. J Med Genet, 2002, 39(12):900-905.( 1) 1) |
| [22] | Liu Y, Zhang H, Zhou K, et al. Tagging SNPs in nonhomologous end-joining pathway genes and risk of glioma[J]. Carcinogenesis, 2007, 28(9):1906-1913.( 1) 1) |
| [23] | Hayden P J, Tewari P, Morris D W, et al. Variation in DNA repair genes XRCC3, XRCC4, XRCC5 and susceptibility to myeloma[J]. Hum Mol Genet, 2007, 16(24):3117-3127.( 1) 1) |
| [24] | Tseng R C, Hsieh F J, Shih C M, et al. Lung cancer susceptibility and prognosis associated with polymorphisms in the non-homologous end-joining pathway genes: a multiple genotype-phenotype study[J]. Cancer, 2009, 115(13):2939-2948.( 1) 1) |
| [25] | Sturgis E M, Zhao C, Zheng R, et al. Radiation response genotype and risk of differentiated thyroid cancer: a case-control analysis[J]. Laryngoseope, 2005, 115(6):938-945.( 1) 1) |
| [26] | Yuan K, Huo M, Sun Y, et al. Association between x-ray repair cross-complementing group 3 (XRCC3) genetic polymorphisms and papillary thyroid cancer susceptibility in a Chinese Han population[J]. Tumour Biol, 2015, DOI:10.1007/s13277-015-3882-4.( 1) 1) |
| [27] | Shen M R, Jones I M, Mohrenweiser H. Nonconservative amino acid substitution variants exist at polymorphic frequency in DNA repair genes in healthy humans[J]. Cancer Res, 1998, 58(4):604-608.( 1) 1) |
| [28] | Bao Y, Jiang L, Zhou J Y, et al. XRCC1 Gene Polymorphisms and the Risk of Differentiated Thyroid Carcinoma (DTC): A Meta-Analysis of Case-Control Studies[J]. PLoSOne, 2013, 8(5):e64851.( 1) 1) |
| [29] | Ryu R A, Tae K, Min H J, et al. XRCC1 polymorphisms and risk of papillary thyroid carcinoma in a Korean sample[J]. J Korean Med Sci, 2011, 26(8):991-995.( 1) 1) |
| [30] | Chiang F Y, Wu C W, Hsiao P J, et al. Association between polymorphisms in DNA base excision repair genes XRCC1, APE1, and ADPRT and differentiated thyroid carcinoma[J]. Clin Cancer Res, 2008, 14(18):5919-5924.( 1) 1) |
| [31] | Zhu Q X, Bian J C, Shen Q, et al. Genetic polymorphisms in X-ray repair cross-complementing gene 1 and susceptibility to papillary thyroid carcinoma[J]. Chin J Epidemiology, 2004, 25(8):702-705.( 1) 1) |
| [32] | Rahimi M, Fayaz S, Fard-Esfahani A, et al. The role of Ile3434Thr XRCC7 gene polymorphism in Differentiated Thyroid Cancer risk in an Iranian population[J]. Iran Biomed J, 2012, 16(4):218-222.( 1) 1) |
 2016, Vol. 30
2016, Vol. 30

